この記事は、GBIFサイエンスレビュー2019にも掲載されています。サイエンスレビューは、研究や政策の分野においてGBIF上のデータが利用された重要で注目に値する事例に焦点を当てています。
作物、家畜、およびその野生近縁種の遺伝子の多様性を維持することは、国連の持続可能な開発目標(SDG)ターゲット2.5、生物多様性愛知目標13、世界植物保全戦略ターゲット9、食糧及び農業のための植物遺伝資源に関する国際条約(ITPGRFA)第5条を含む複数の国際協定で優先事項とされています。これらの目標に対する進捗状況を測定できるように、数々の指標が提案されたり、展開されたりしています。
コロンビアの国際熱帯農業センター(CIAT)のColin Khouryは、これらの指標の1つの開発に関与してきました。
「既存の指標について調べてみると、方法論とデータの両方に不備がありました。その多くは動かぬ数字を示していましたが、それが十分かどうかを示すものはありませんでした。数字からは多くが伝わらず、いつが完了なのかもわかりません。」
Khouryは、農業従事者としての実践的な農業に終わりを告げ、研究の世界に戻って農業の多様性の保全に関する研究に携わり始めました。彼は博士課程の学生のとき、豆類とキャッサバの世界的なコレクションの拠点であるCIATの種子バンクで時間を費やし、種のオカレンスデータのモデリングに基づくギャップ分析の方法論を開発しました。これは、CIATと他の種子バンクがコレクションの網羅性を究明するのに役立ちました。
「我々は妥当な結果を得てプロジェクトを終了しましたが、ギャップ分析にはさらなるポテンシャルがあると思いました。2017年に、生物多様性指標パートナーシップが目標13の指標の追加を呼びかけました。我々は我々の方法論を提案し、指標を開発する9カ月間のプロジェクトの研究助成を受けました。」
Ecological Indicators誌の論文に詳述されているように、提案された方法論にはオープンデータにすべて依存した、シンプルで再現可能な手順が含まれていました。まず、GBIFと種子バンクのデータベースからオカレンスレコードをダウンロードし、関連する種の動的リストを作成します。これらを気候の共変量と組み合わせて使用し、全種の潜在的分布をモデル化します。既存の遺伝資源コレクションにおけるこの分布と指定されている保護地域とを比較することにより、保護スコアを導き出します。このスコアから国家、地域、地球規模の指標を計算することができます。
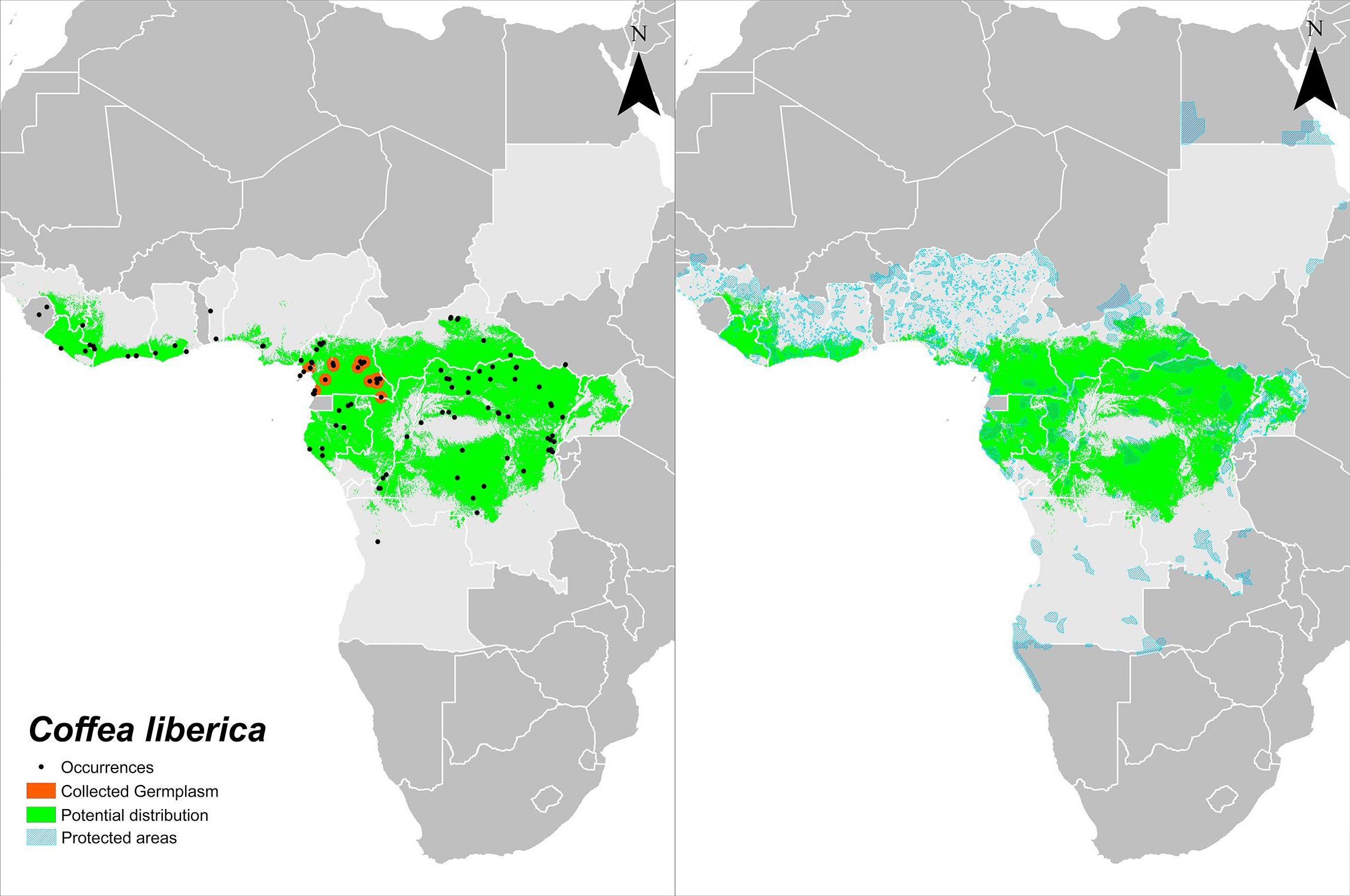
オカレンスの場所に基づくCoffea libericaの潜在分布モデル。淡色背景は、モデルをクリップするのに用いられた在来国境を示す。潜在分布モデル上で、約50kmの緩衝地帯(赤)により囲まれている「遺伝資源が収集されたサイト」以外の地点。指定保護区域の内外で発生しうる潜在分布。図はKhouryら(2019) (CC BY 4.0)を改変。
近年、世界自然保護区データベースが更新されたことを受け、Khouryと同僚らは再度解析を行い、その指標が公式に承認されました。
「これはエキサイティングなニュースです!我々の指標が生物多様性指標パートナーシップのウェブサイトに掲載されます。このウェブサイトは、国家代表がこれらのターゲットに関する進捗状況を報告するためのリソースとなるものです。我々の場合は愛知目標13です。」
Khouryとそのチームは、完全な開示性と透明性をもたせるために、ほぼ同時期に出版された データペーパーの一部として論文中で編集、生成された全データを公開しました。また、オンラインで結果を調べられるインタラクティブ版も作成しました。 こうしたアクセスしやすい情報へのこだわりは、Khouryと彼の所属機関がオープンデータについていかに熱意を持っているかを示しています。また、彼はGBIFなしでは指標を実現できなかったと完全に認めています。
「私はこの仕事を始めた頃、農業の種子と多様性に心を奪われました。それ以来、私は次の3つのことに焦点を当て続けてきました:それをどのように処理するか、良い目的のためにどのように使うか、そして最後に、だれもができるだけ多くアクセスできるようにすることでどのように関心を高めるかという3点です。」
彼はこう結論付けています。「GBIFは、我々が生物の多様性について理解する方法の1つです。それについての情報にアクセスできないとしたら、どうやってそれを保護することができるでしょうか。」